寻找好牛有方法!
更好的牛

利用16S rRNA扩增子与宏基因组测序探究牛胃肠道微生物与甲烷排放的关系
近来肠道菌群的研究简直要逆天,不仅跟肥胖及诸多慢性病有密切关联1,
能用来预测抗免疫抑制疗法的疗效2,还可以迁徙到肠道以外的肝脏3!

看来人类健康管理的要诀之一就是管理好你身上的第二个基因组—肠道菌!
原来,古代尝粪忧心的孝子庾黔娄4和表忠心的越王勾践5,很早就明白了这个道理啊。

那么,纵横动植物界,略懂医疗与畜牧的大阅哥就好奇了。
肠道菌群研究除了具备医学应用价值之外,在畜牧业是否也有实际的应用呢?
【除了汽车尾气和氟利昂之外,畜牧业也是地球暖化的元凶之一】
全球饲养的牛只约有10亿头(时间简史上说15亿头)。牛在消化或反刍过程中产生的屁等排泄物会产出100多种污染气体,其中就包括了大量的甲烷。甲烷属于温室气体,对地球暖化的影响比二氧化碳高25倍以上。每头牛每天的甲烷排放量约在200-280升(约140-200克),畜牧业甲烷排放量占全球甲烷排放总量1/3(大家可以算一下10亿 x 200升是多少)。而全球由牛排放的二氧化碳更是占全球温室气体总排放量的18%,甚至超越了人类交通工具,如汽车、飞机。2014年,美国政府曾经对奶牛展开治理行动,协助屠宰场、奶牛场和大型牧场建沼气池,并且鼓励由伊利诺斯乳业创新中心提出的“未来之牛”计划,该计划的目标是通过选择性育种培育出能够防止产生甲烷的谷物。希望“饲养的奶牛可以产更多的奶,变得更健康,同时还可以解决排放甲烷的问题。”

【利用调控肠道菌来管理牛放屁】
甲烷不会被牛自身利用,反而会造成能量流失和对环境造成了不良影响。影响甲烷产生的一个重要因素就是牛胃中微生物多样性的不同,要对他们进行研究,就该扩增子与宏基因组测序登场啦。

牛瘤胃中的微生物组成与甲烷排放量密切相关,但人类对所参与的蛋白了解很少。
2015年,英国阿伯丁大学的团队从72只肉牛中选取了4对甲烷排放量超级高和超级低的,记录了它们的饮食记录。研究团队利用qPCR的方法对微生物的16S/18S rRNA 基因对菌群结构做了大致评估,并对样本中微生物的宏基因组进行了深度测序和拼接。6
16S/18S rRNA基因qPCR结果显示,甲烷短杆菌属(Methanobrevibacter)在产甲烷最高的牛胃中高出2.5倍,而琥珀酸弧菌科(Succinivibrionaceae)在高产甲烷的牛胃中要比低产甲烷牛胃中低上4倍。
此外,研究人员通过对宏基因组测序数据的拼接找到了150万个预测蛋白,其中只有不到一半(48.6%)有Pfam domain注释信息。接下来比对KEGG数据库,在20个与甲烷排放相关的直系同源组(KO,KEGG orthology)比对出所鉴定到的2,774预测的蛋白,这些蛋白可以区分甲烷高产和低产的牛只(下图),但是仅有16个(0.6%)在已知数据库中有100%的匹配。本研究显示菌种基因丰度可以作为甲烷排放的研究指标,但对相关基因或蛋白的功能尚待进一步探索。
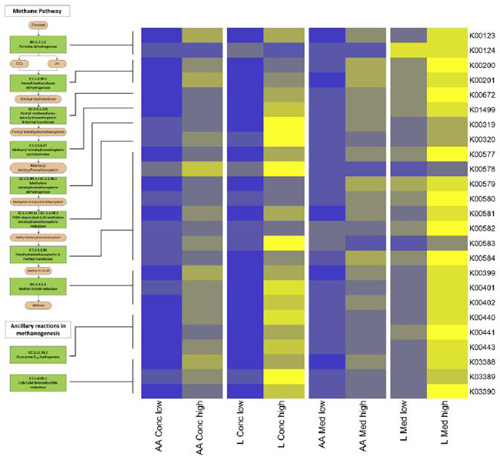
8个样本(4个对应组)的不同蛋白丰度聚类热图(KO代表与甲烷生成相关的直系同源组)
牛瘤胃中甲烷短杆菌属的菌种分布影响甲烷产生量
17年2月,瑞典农业科学大学的研究团队的团队对21只分别在三个月的相同喂养条件下,持续低,中,高甲烷排放量的奶牛(12 只荷斯坦牛和 9 只瑞典红牛)在纤维消化,发酵终产物和瘤胃中细菌、古生菌的组成上进行研究。研究方法分为对牛瘤胃中菌群进行16S rRNA V4区测序及对甲烷短杆菌属(Methanobrevibacter)菌群体进行荧光定量分析两部分。结果显示,虽然甲烷短杆菌属(Methanobrevibacter)在甲烷高、中、低产的牛之间没有显著差异(只有趋势),但该属中的单胞菌种Methanobrevibacter ruminantium 如果占比越多,则牛屁中的甲烷含量就越低。7
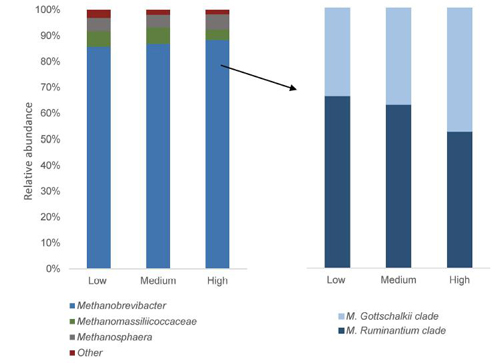
左图代表菌属组成(蓝色为甲烷短杆菌属Methanobrevibacter),
基于16S rRNA扩增子测序的结果,基于序列结果OTU 水平上的主坐标分析 (PCoA) 发现了两个差距较大的簇,分别对应高甲烷产出和低甲烷产出(中等甲烷产生在两个簇里面都存在)。这两个簇的主要区别在于普氏菌属Prevotella的丰度不同,此外,和英国科学家的发现一致,低甲烷产出的簇中有较多的序列属于琥珀酸弧菌科Succinivibrionaceae家族。研究也进一步讨论了低甲烷产出的奶牛可以更加有效率的利用饲料与牛奶产量(在低甲烷产出中丙酸盐比例较高而高甲烷产生中丁酸盐比例较高),可能源自于微生物群体和优势菌种基因功能的不同。

样本OTU水平的主坐标分析图:绿色点为低甲烷产出组,红色为高甲烷产出组,黑色为中等甲烷产出组。
科学家除了想要找到放屁更环保的牛之外,也用尽办法在找更高产的牛。有研究指出8,9,荷斯坦牛的β-酪蛋白编码基因CSN2的A2型,对于奶蛋白含量和产奶量较A1型有显著提高,且能避免A1型β-酪蛋白的人体消化水解代谢产物(BCM-7)所造成的相关疾病。目前最主要的奶牛品种荷斯坦牛中,A2A2型奶牛的比例仅占不到30%,因此国内外已经开展基因优选奶牛的牧场,也将提升A2型奶牛基因比例作为重点工作之一。
阅微基因不仅可以提供人肠道菌群分析,也可以提供牛的胃肠道菌群分析服务,以及牛β-酪蛋白分型服务。
请关注我们,我们也将与您在“2018(第四届)肠道微生态与健康国际研讨会”(3月30-31,上海)
和中国肠道大会(5月11-13,北京)相见噢!
参考文献:
2. Gopalakrishnan, V., et al. "Gut microbiome modulates response to anti–PD-1 immunotherapy in melanoma patients." Science (2018): 97-103.
3. Vieira, S. Manfredo, et al. "Translocation of a gut pathobiont drives autoimmunity in mice and humans." Science (2018): 1156-1161.
4. 二十四孝。
5. 《吴越春秋》。
6. Wallace, R. John, et al. "The rumen microbial metagenome associated with high methane production incattle." BMC genomics (2015): 839.
7. Danielsson, Rebecca, et al. "Methane production in dairy cows correlates with rumen methanogenic and bacterial community structure." Frontiers in microbiology (2017): 226.
8. Thakur, Keshav, and Akshay Anand. "Milk metabolites and neurodegeneration: Is there crosstalk?." Annals of neurosciences (2015): 239.
9. 薛杰, 苗勇旺 家畜生态学报 第36卷第10期(2015)牛科物种β-酪蛋白基因的研究进展。






