先看看问题背景
无论是科研用细胞系在文章发表前的鉴定,还是在生物制药和细胞治疗中的研发/生产细胞质控,细胞STR鉴定都是一项常规但很重要的工作。
我们都知道,细胞系的STR分型被检测出来后,通常需去ATCC或DSMZ数据库进行8个核心STR位点+ 1个Amelogenin位点的比对,以得到匹配结果与对应匹配百分比,从而得以知晓细胞系身份。根据ANSI 制定的算法,当细胞系的匹配度≥80% 时,认为它们具有相关性,即衍生于共同的祖先细胞;匹配度在56%-79% 之间,需要进一步验证相关性;小于56%,表明两者不具有相关性。
在实际情况下,细胞系由于连续培养传代,常常会发生基因变异,例如发生微卫星不稳定、杂合性缺失、染色体数目变化等情况。也可能会发生无法去除的细胞污染,例如Chang liver cell已经证实被HeLa cell污染。在这种情况之下,此国际通用,且沿用了多年的9位点比对方法可能不足以对细胞系身份进行准确鉴定。那么如果加测位点数量,是否能够提高鉴定的准确性和区分度呢?让我们从武汉大学研究团队近期发表的一篇文献中来一探究竟吧。
让实验数据来回答我们的疑问
21位点的细胞STR鉴定在检测人类细胞系交叉污染中的优势
Advantages of a 21-loci short tandem repeat method for detection of cross-contamination in human cell lines
武汉大学生命科学系、药学院,武汉大学中国典型培养物保藏中心
实验设计与研究方法
该研究选取了197株不同的细胞系和299个无关个体样本进行21个STR基因位点的检测,利用ATCC的算法(待测细胞株所有匹配峰个数∕ATCC数据库中对应细胞系所有峰的个数)分别在使用9个STR位点(包含在21位点中)和21个STR位点的情况下进行不同样本之间的两两比对,然后计算每个配对间的匹配度(细胞系样本和无关个体样本共有19,306个和44,551个配对)。
① 使用21位点检测显著提升鉴定准确性与“不确定样本”区分度
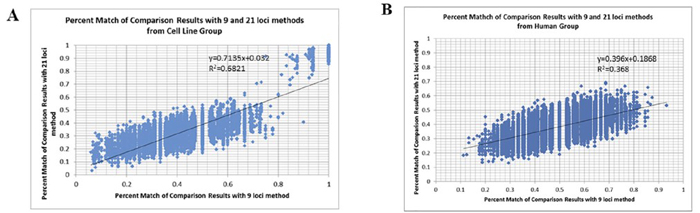
分别对细胞系和无关个体的匹配度数据(使用9位点和21位点)进行回归统计分析,得到的回归系数分别为R2= 0.6821(细胞系)和R2= 0.368(无关个体)。推测无关个体回归系数较低可能是由于在细胞系样本中部分细胞系来源相近或相同,但无关个体的基因背景差异较大而导致的。研究者选取无关个体样本进行分析,目的应该是为了与细胞系样本进行对比研究,探究不同位点数量在区分遗传背景差异较大的样本上的表现。
图1.细胞系样本(图A)和无关个体(图B)中使用9位点(X轴) & 21位点(Y轴)配对匹配度回归分析
接着,对不同组间配对的匹配度进行详细分析。使用9位点计算时,细胞系样本中“需进一步验证相关性”(Uncertain authentications,匹配度 55%-80%;值得注意的是:在ATCC数据库比对时,该匹配度区间应该为 56%-79%)的比对结果所占比例为8.65%,当位点增至21个时,比例下降到0.22%。该比例在无关个体中也随着位点增多,从29.11%降低到0.67%。两种不同样本的比例平均降幅高达97.50%。该分析结果显示在利用21个位点计算时匹配度时,显著降低了细胞系鉴定中的不确定性,提升了鉴定结果的准确性。
表1.细胞系与无关个体样本中不同比对匹配度的个数和所占比例详细情况
图2. 细胞系样本中9位点/21位点不同比对匹配度所占比例
黑色柱代表9位点,白色柱代表21位点。(红色框中为匹配度55-80%区间的对比)
② 使用21位点检测可提升对来源接近的细胞系的分辨率
在细胞系样本的鉴定中,使用9位点计算时,共有611个细胞系被鉴定为“具有相关性,衍生于共同的祖先细胞”(Related authentications, 匹配度 ≥ 80%),当使用21个位点时,该数量为589个。虽然两种计算方法的计算结果只差了13株细胞,但通过对这13株细胞的差异进行研究发现21个位点可以提供更加可靠的结果,即便两株细胞的位点信息是十分接近的。
例如,前文提到的HeLa cells 和 Chang liver cells 细胞系中,在使用9位点计算时,匹配度为93%,而使用21位点计算时,匹配度就降为77.33%了。更有趣的是下表中的三对细胞系 (CAL 27 and SiHa-1, Caco-2 and HCC827, Caco-2–2 and HCC827),当使用9位点计算时,匹配度分别为81.82%, 80%, and 80%, 然而当位点增到21个时,其匹配度下降至47.62%,44.07%和44.83%,可见检测结果的差异之大,有的细胞系在9个位点之外的位点甚至完全不相符。该结果说明通过增加检测位点,可以更好的提升来源相近细胞系的分辨率。
表2 分别使用9位点和21位点计算的“具有相关性,衍生于共同的祖先细胞”匹配度
所以我们该怎么做?
虽然目前在ATCC和DSMZ这种国际主流的数据库里依然要求用9个位点进行比对,但该种比对方法确实在一些情况之下无法对细胞系的身份做出准确鉴定。本篇文章利用大量样本进行多种分析得出,相较于只比较9个位点,检测21个位点可以显著的提升检测的准确性,降低检测中“不确定相关性”的比例,同时能够更准确的区分来源相近的细胞系。
在实际涉及到细胞系使用的科研和生产工作中,我们强烈建议使用稳定、可靠、位点数目合适的检测方法,从而对细胞系进行更加准确的鉴定,尤其在以下情况中:
场景一:自建细胞系时,可以得到和保存更多细胞位点的信息。
场景二:对细胞比对结果有疑问,需要进一步确认时。
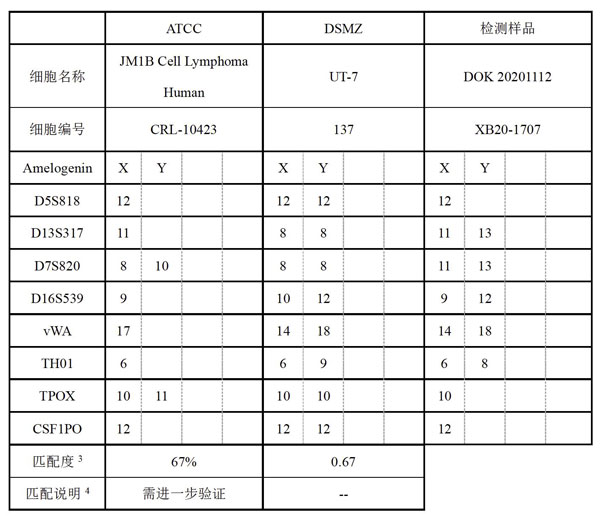
在对ATCC或DSMZ比对结果存在疑问时,如何用多检测了的位点来确认到细胞信息呢?这时可以利用其他数据库进行辅助比对,在这里我们拿一株名称为“DOK 20201112”的客户送检细胞来举例。该株细胞在ATCC和DSMZ数据库比对结果如下图所示:
图3. 细胞系与ATCC和DSMZ数据库比对结果
通过比对,可得知该细胞在ATCC数据库中最高匹配度细胞为“JM1B Cell Lymphoma Human”,匹配度为67%,匹配说明为“需要进一步验证”,在DSMZ中匹配细胞为“UT-7”,匹配度为0.67。
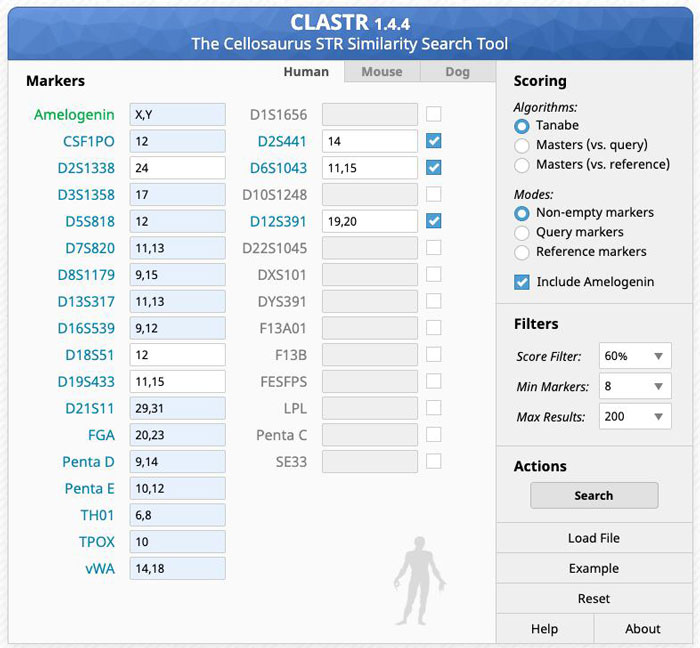
为了进一步验证该细胞,我们利用ExPASy Cellosaurus数据库进行检索,该数据库可以在检测时输入多个STR位点信息用于比对。如下图所示:
图4. 细胞系在ExPASy Cellosaurus数据库中利用21个位点信息进行检索
通过比对,由下图我们可以看到,匹配度结果最高的细胞为“DOK”细胞,待测细胞与数据库中比对细胞共有位点数为16个,匹配度为98.18%。这与客户送检细胞的名称是一致的。
图5. 待测细胞系在ExPASy Cellosaurus数据库比对结果中匹配度最高的细胞位点匹配情况
当然,除了在多个数据库上进行比对、分析。还可以根据相关文献查证的方法来确定待测细胞系的身份。
场景三:在进行细胞质控时,需要进行细胞与细胞之前相互比对的时候,可以等到更加准确的比对结果。
正如上文所述,本篇研究中细胞之间的比对是通过两两比对实现的。我们用文中CAL27和到SiHa-1两株细胞的比对来举例:
注:红框为ATCC数据库中要求的9个位点
在此比对中,将两株细胞当中其中的一株作为“待测细胞株”(SiHa-1),另一株当做“被比对的细胞株(CAL27)” 。在用21位点进行比对时,用SiHa-1细胞的和比对细胞(CAL21)的匹配峰个数(15个)/SiHa-1细胞总峰个数(29个),得匹配度为51.72%。而在使用9位点比对时,匹配度为81.82。这说明试用21位点检测时,显著提升了细胞两两比对时的鉴定准确性和区分度。
各位老师就可以用这个方法来比对两株细胞,进行核查验证啦。
值得一提的是,本篇文章当中所使用的检测体系就是阅微基因细胞STR鉴定的检测体系哦,阅微基因的细胞鉴定服务目前已完成2万余株细胞系的检测,与上百家科研单位、细胞治疗、基因治疗、生物药厂、疫苗厂和细胞库等机构长期稳定合作,深受客户认可。
总之,细胞STR鉴定,就找阅微基因!